Heute machen wir einen Ausflug in mein Computermuseum. Ich habe nämlich die Kurzanleitung von 1993 zu der Retrieval-Sprache grips vom DIMDI wiedergefunden. Ich war schließlich schon 1988 ein Fan von grips und Medline. Mir stellt sich die Frage, ob die heute so beliebte AI Suche mit ChatGPT besser ist als die mit grips beim DIMDI vor 36 Jahren. Was die Ergebnisse anbetrifft.
Frühere Posts zu medizinischen Internetrecherche
Medline mit dem Akustikkoppler
Evidenzbasierte Medizin
Medizinische Internetrecherche
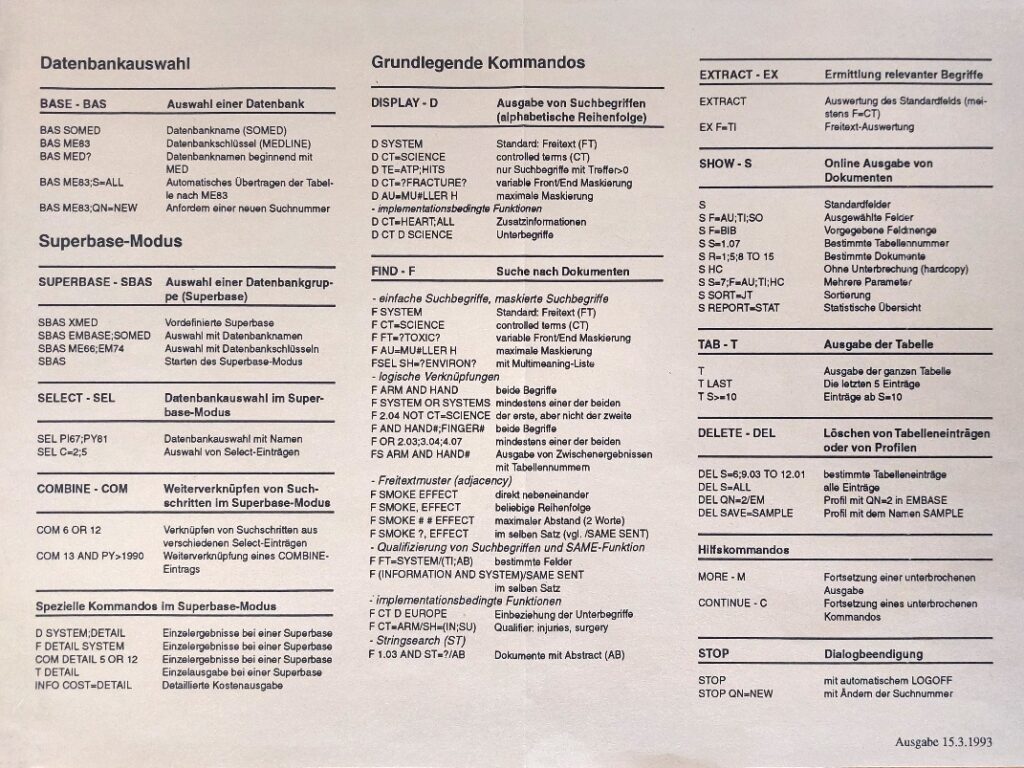
Kurzanleitung von 1993 zu der Retrieval-Sprache grips vom DIMDI

Zur Geschichte des DIMDI
Das DIMDI in Köln hätte die NLM Deutschlands werden können. Im Jahr 2020 ist das DIMDI dann in das BfArM eingegliedert worden. Hier der Link beim BfArM zur Geschichte des DIMDI, hier ein Text über das DIMDI beim Ärzteblatt. Im Prinzip war das DIMDI ein spezialisierter IT-Dienstleister im Gesundheitswesen, mit eigenem Rechenzentrum und der selbst entwickelten, rein textbasierten Retrieval-Sprache grips. Die war schon 1975 online verfügbar, also lange vor dem heutigen Internet. 1997 erfolgte der Release von grips-WebSearch. Später kamen die Verwaltung und Entwicklung von Klassifikationssystemen als Aufgaben hinzu. Mit der freien Verfügbarkeit von Medline über die NLM endete für mich die Nutzung des DIMDI.
Dialog mit grips bei DIMDI
| BAS ME83 | Datenbankauswahl Medline ab 1983 |
| F suchbegriff | Finde suchbegriff |
| EX F=CT | Erzeuge eine Liste der MESH-CT nach Häufigkeiten |
| F 3.04 AND PY>1990 | Wähle ein Listenelement aus und begrenze das Publikationsdatum |
| S F=AU;TI;SO;AB | Gib die Datenbankfelder Autor, Titel, Quelle und Abstract aus. |
| INFO COST=SESSION | Was hat die Sitzung gekostet? |
Das ist ein Beispiel einer extrem geradlinigen Suche bei Medline auf DIMDI. Die war in kürzester Zeit durchgeführt. Durch die EX-Funktion konnten die wesentlichen MESH-Begriffe gefunden werden. Auch eine exploding search war mit den CTs möglich, also die Berücksichtigung der Unterbegriffe der CTs. Das Ergebnis per Mitschnitt der Sitzung war eine reine Textdatei, die weiter bearbeitet werden konnte. So einfach ist das, wenn die Daten von einer hochstrukturierten, indexierten Quelle stammen. Die Kosten lagen vielleicht bei 50 DM. Also für Privatpersonen recht teuer.
Vergleich mit einem AI Search String
Wie performt im Vergleich dazu die künstliche Intelligenz? Ich habe den komplexen Search String aus einem AutoWriter Plugin for WordPress in nichtmedizinischem Umfeld getestet. Hier ist er, ich habe ihn für die Übersichtlichkeit gegliedert. Es ist gut erkennbar, dass der eigentlichen Fragestellung nur sehr geringe Aufmerksamkeit zukommt: „Create a compelling and well-researched article of at least 500 words on the topic of “carbon mineralization” in English.“ Das Hauptaugenmerk liegt somit auf formalen Aspekten der Präsentation. Hier das Ergebnis bei der Anwendung von GPT-4.
Das mit dem AI Search String ist ja ganz gut gedacht. AutoWriter klingt auch gut. Aber worauf bezieht sich die AI denn? Was sind ihre Quellen? Wie kann ich Einfluss nehmen auf die stufenweise Entwicklung der Fragestellung? Ich finde die Tatsache, dass die AI jegliche Datenfelder der Quellen zusammenfasst, ein Unding. Für hoch spezialisierte Suchen ist das völlig unzureichend, wenn die Ergebnisse verlässlich sein sollen. Das obige Ergebnis wäre vielleicht für eine populäre Zeitschrift mit unkritischen Lesern geeignet.
Wichtig: Von künstlicher Intelligenz würde ich schon rein semantisch erwarten, dass diese unerwartete Zusammenhänge erkennen und damit echte neue Erkenntnisse liefern könnte. Sorry, es tut mir leid, schön labern können ist heutzutage bestimmt zeitgemäß, hat aber mit Wissenschaft und Evidenz noch nichts zu tun.
Zusammenfassung
Ach ja, die heutige AI Suche ist fachlich gesehen keinesfalls besser als die historische Suche mit der extrem kompakten Retrieval-Sprache grips bei DIMDI. Die Ergebnisse waren damals viel spezifischer als bei der Anwendung von AI. Wer es wirklich genau wissen will, sollte im medizinischen Umfeld die Advanced Version von PubMed nutzen. Bisher habe ich dort die EX oder EXTRACT function wie bei grips noch nicht gefunden. Eigentlich bin ich ja auch im Ruhstand. ;<D